
1.基于质谱的肽段鉴定方法可以更直接、准确地鉴定到免疫肽;
2.质谱数据de novo从头测序技术,不依赖于已有数据库,可以发现更多的非典型免疫肽;
3.可以分析NGS技术无法获得的免疫肽潜在翻译后修饰信息;
4.特有的AI算法,不依赖于个性化测序,提高了免疫肽组分析的效率;
5.领先的高分辨质谱仪;
6.专业的技术人才;
免疫肽组是指对有核细胞在参与抗原呈递途径中的 MHC I 类或 II 类蛋白质(Major Histocompatibility Complex,也叫主要组织相容性复合体,如人类 MHC 的表达产物 HLA,Human Leukocyte Antigen)上呈递的全部多肽的研究,对于探究细胞介导和体液反应介导的免疫原性表位至关重要 [1]。
免疫肽组分析是对 MHC I 类和 MHC II 类免疫肽的定性和定量分析,不仅能够发现个性化癌症免疫疗法的靶点(例如来自肿瘤特异性突变的新抗原或肿瘤相关抗原),还有助于开发新的mRNA / 多肽疫苗和免疫细胞疗法[2]。近年来, 随着质谱技术的革新和细胞免疫疗法的进展也大力加速了免疫肽组学的发展。
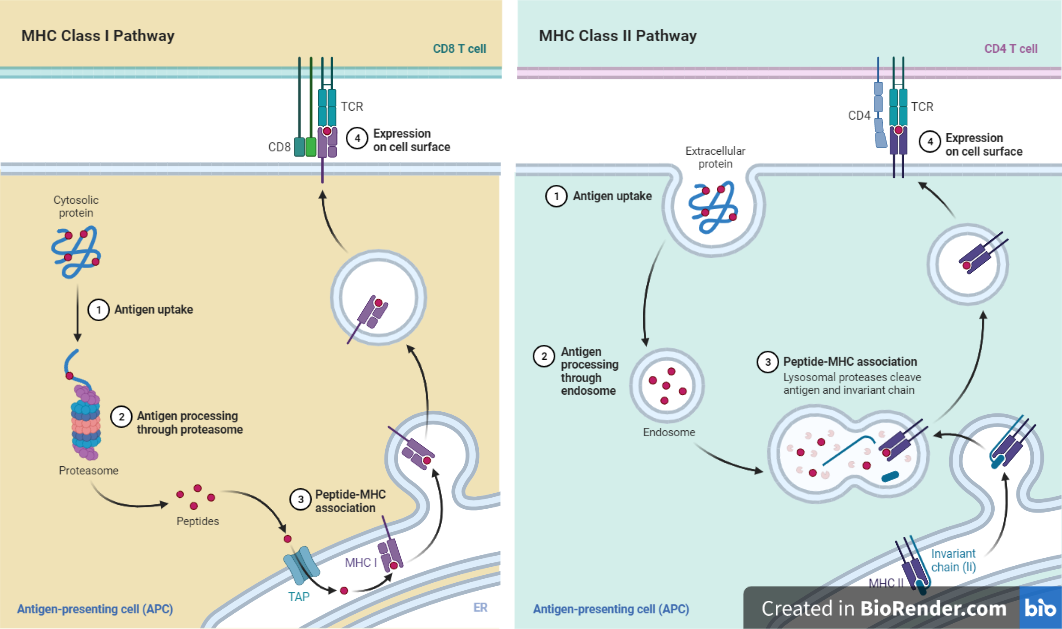
图 | HLA 抗原呈递过程(引自 bioRender 网站制作)
DeepImmu 是以个性化DeepNovo算法为核心的独有的肿瘤新抗原分析平台,从蛋白质组学质谱技术出发,结合快速、精确的 DeepNovo 算法,使用高灵敏度质谱仪,加速免疫治疗的研发进程。
作为多肽 / 蛋白从头测序行业先驱者和金标准,DeepNovo算法是 BSI 研究团队在 2017 年发表的世界首个应用深度学习对多肽序列从头预测的算法 [3],并且在过去近 4 年的时间,该算法也从 DDA 的采集模式,到对 DIA 数据采集模式首创从头测序 [4],再到加入离子淌度维度信息和摆脱对质谱仪的质量精度依赖的改进算法[5]。DeepImmuTM是以个性化DeepNovoTM算法为核心的独有的肿瘤新抗原分析平台,个性化深度学习技术提高了从头测序的精度 [2]。
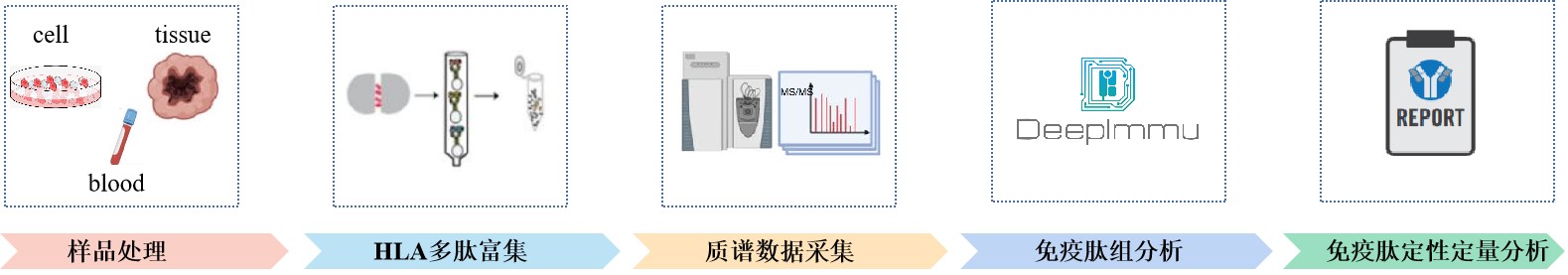
百蓁生物结合高效特异的免疫肽富集技术、先进的质谱仪器和 DeepImmuTM技术平台为客户提供一站式免疫肽组分析服务解决方案,可根据客户的研究目的以及样本特点,进行定制化实验设计。DeepImmuTM技术平台在降低了样品量的同时能够可重复地鉴定并定量出尽可能多的免疫肽。
1.基于质谱的肽段鉴定方法可以更直接、准确地鉴定到免疫肽
2.质谱数据de novo从头测序技术,不依赖于已有数据库,可以发现更多的非典型免疫肽
3.可以分析NGS技术无法获得的免疫肽潜在翻译后修饰信息
4.特有的AI算法,不依赖于个性化测序,提高了免疫肽组分析的效率。
5.领先的高分辨质谱仪
6.专业的技术人才
7.高效稳定的实验平台,完整的试验记录,确保试验结果的准确以及可追踪
8.严格的质控
样本类型 | 细胞 | 组织 | 血液 |
样本最低需求量 | 107 | 50mg | 全血:10ml 血浆:5ml 血清:5ml |
样本制备要求 | 细胞样品需保持90%以上的活细胞率,同时防止支原体污染。 | 新鲜制备是为最佳;液氮速冻保存。注:防止血液污染 | 新鲜制备是为最佳;液氮速冻保存。注:防止出现溶血现象 |
样本运输要求 | 使用足量干冰运输,期间避免冻融,尽量选用顺丰快递,防止样品降解。 | ||
交付标准 (报告内容) | 1.免疫肽定性结果以及对应的蛋白信息列表 2.免疫肽定量结果列表 3.试验过程中QC结果 4.免疫肽长度分布展示图 5.免疫肽聚类分析 6.免疫肽定量结果统计分析 7.免疫肽对应蛋白功能富集分析 8.客户特殊分析需求结果 9.完整的实验报告 | ||
2021 年来自洛桑大学的HuiSong Pak[6]等研究人员在 MCP 发表的题为“Sensitive Immunopeptidomics by Leveraging Available Large-Scale Multi-HLA Spectral Libraries, Data-Independent Acquisition,and MS/MS Prediction”一文中,使用不同的建库策略进行免疫肽组的DIA 分析检测,文中所使用的分析平台为maxquant(用来建库)结合Spectronaut(用于DIA 分析)软件,为了说明 DeepImmu 平台的优势,我们使用 DeepImmu 平台,使用两种不同的分析策略 library based search 和database based search 对文献中的数据进行了重新分析,结果表明不管是 library based search 和 database based search 的方法,均大幅提高了免疫肽的鉴定数目,并且文中鉴定到的免疫肽也绝大多数在我们鉴定到的结果中。同时database based search 鉴定的数目还多于 library based search 鉴定到的(见下图),这也为我们做DIA分析减少了建库的流程。这表明使用DeepImmu 平台可以提高免疫肽鉴定数目,同时简化试验流程。
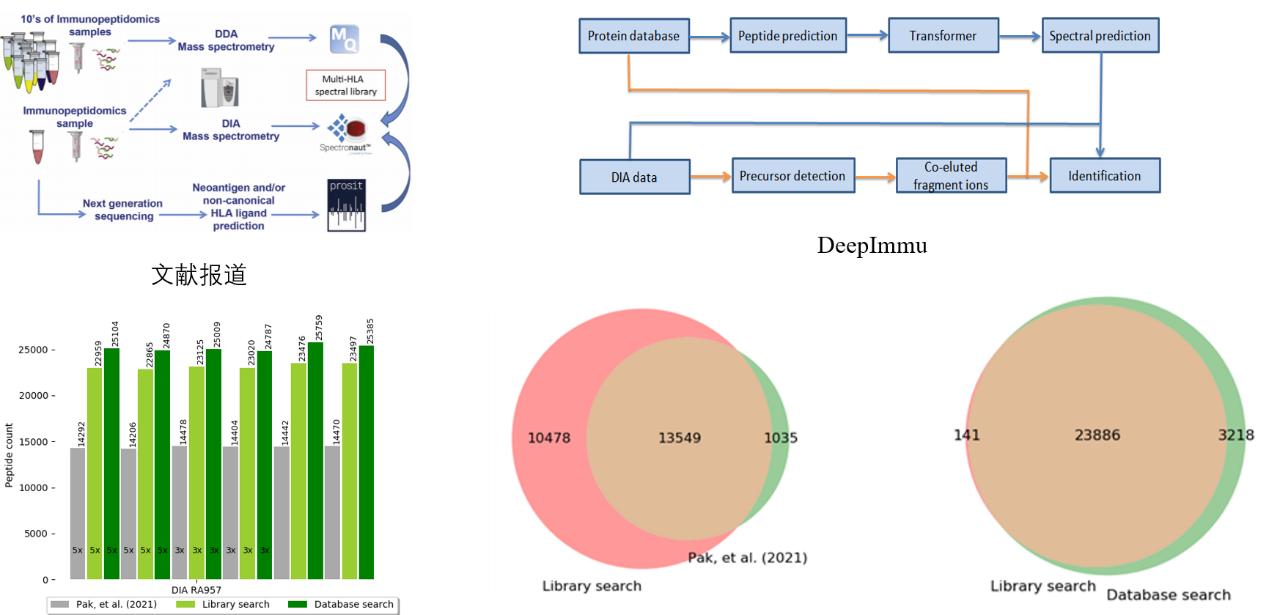
图 | DeepImmu 重新分析 Pak et al 结果
1. Chong C, Coukos G, Bassani-Sternberg M. Identification of tumor antigens with immunopeptidomics [J]. Nat Biotechnol, 2022, 40(2): 175-188.DOI: 10.1038/s41587-02101038-8.”
2. Tran NH, Qiao R, Xin L, et al. Personalized deep learning of individual immunopeptidomes to identify neoantigens for cancer vaccines [J]. Nature Machine Intelligence,2020, 2(12): 764-771.DOI: 10.1038/s42256-020-00260-4.”
3. Tran NH, Qiao R, Xin L, et al. Deep learning enables de novo peptide sequencing from data-independent-acquisition mass spectrometry [J].Nat Methods, 2019, 16(1): 6366.DOI: 10.1038/s41592-018-0260-3.”
4. Tran NH, Zhang X, Xin L, et al. De novo peptide sequencing by deep learning [J]. Proc Natl Acad Sci U S A, 2017, 114(31): 8247-8252.DOI:10.1073/pnas.1705691114.”
5. Qiao R, Tran NH, Xin L, et al. Computationally instrument-resolution-independent de novo peptide sequencing for high-resolution devices [J]. Nature Machine Intelligence,2021, 3(5): 420-425.DOI: 10.1038/s42256-021-00304-3.”
6. Pak H, Michaux J, Huber F, et al. Sensitive Immunopeptidomics by Leveraging Available Large-Scale Multi-HLA Spectral Libraries, Data- Independent Acquisition, and MS/MSPrediction [J]. Mol Cell Proteomics, 2021, 20(100080.DOI: 10.1016/j.mcpro.2021.100080.”