来源:百小蓁发布时间:2023-11-01浏览量:108次
PEAKS用户顶刊好文推荐: 2023年10月19日,来自全球第二大生物技术公司Genentech的研究团队,在Nature Biotechnology杂志上发表了一篇题为 Systematic discovery of neoepitope–HLA pairs for neoantigens shared among patients and tumor types的文章,他们发掘了86个具有免疫原性的候选新生抗原-HLA结合表位,本研究的相关数据将非常有助于研究人员推动在抗原加工以及新抗原表位靶向治疗方面的研究进展。本研究使用了PEAKS Online软件完成了对工程T细胞系的全免疫多肽组质谱数据的准确、无偏鉴定。

T细胞在消除癌细胞方面发挥着关键作用1,2,增强内源性肿瘤特异性T细胞活性(例如癌症疫苗)或引入靶向新抗原的T细胞的免疫疗法具有很好的临床疗效3,4。新抗原导向的治疗方法需要肿瘤细胞表面具有可被HLA呈递的新抗原表位,即由突变蛋白衍生的8 – 11个氨基酸多肽。T细胞受体(TCRs)在HLA复合物的背景下与同源肽相互作用,使得治疗靶点既可以是新抗原表位序列又可以针对不同HLA的分型。尽管临床前景看好,但新抗原特异性治疗的应用仍受到限制,尤其是可能是某些癌症亚型的患者中共有的那些,被验证新抗原表位与HLA的结合的范围是有限的。
1. 高通量TR-FRET测定和NetMHCpan-4.0预测结合用于共有新抗原-HLA结合表位的发掘
首先,通过临床基因组学筛选出与36个候选新生抗原结合的16个HLA分型,然后结合时间分辨荧光共振能量转移实验(TR-FRET)和NetMHCpan-4.0的预测功能对候选的抗原结合表位与HLA-多肽复合物进行表征,结果发现两种方法约有30%左右的一致性,且各有优势,在实际研究中,可以结合这两种方法进行新抗原表位的全面发掘。
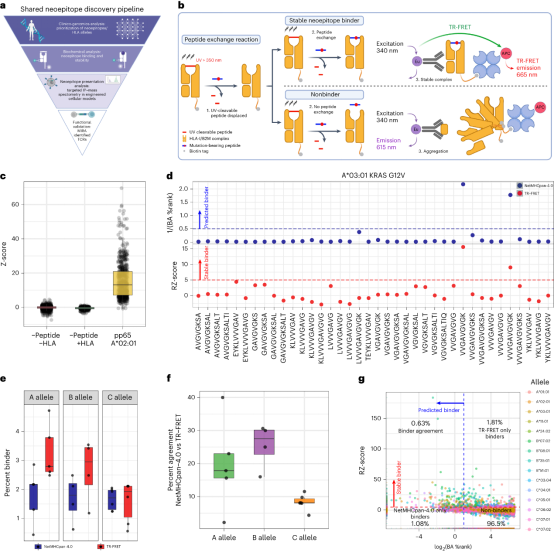
图1 高通量筛选新抗原表位的工作流程
2. 构建稳定表达47种癌症共有新生抗原的I型HLA单等位基因细胞系
选择HMy2.C1R (C1R) 淋巴母细胞,利用CRISPER-Cas9破坏其HLA-C等位基因(HLA-C*04:01),生成HLA-null细胞群,并通过FACS将目标细胞群富集出来。随后,通过稳定的慢病毒转导将15个HLA等位基因作为单个转基因分别引入新抗原连接子表达和非连接子表达的C1R-HLAnull细胞系,最终共获得30个细胞群。连接子和无连接子的新抗原工程细胞构建时包含了一组相同的7个已知的HLA-A*02:01递呈表位。并且,随后的HLA-IP和靶向质谱分析证实了连接子和非连接子HLA-A*02:01工程细胞中存在的两种对照肽和前序报道的TP53 R175H23新抗原表位。
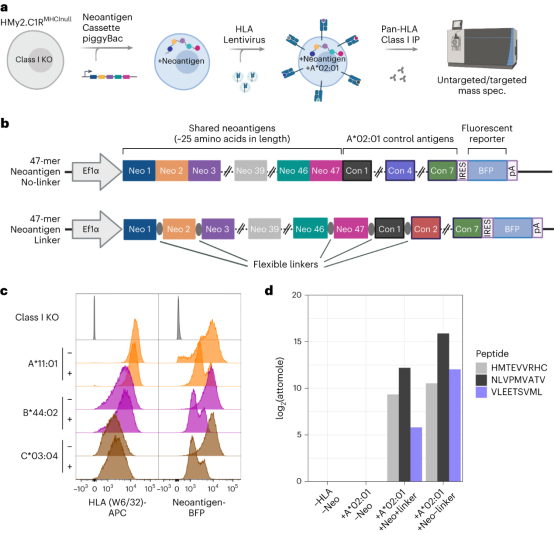
图2 单等位基因工程细胞系的构建
3. 利用质谱技术鉴定单等位基因工程T细胞中的免疫多肽组
工程T细胞构建成功后,通过IP富集出I类HLA多肽,然后通过质谱进行DDA鉴定,得到的质谱原始数据通过PEAKS Online软件完成分析,最终在各个细胞群中分别鉴定出218-6663个特异的长度在8-11个氨基酸的肽段,且每个等位基因对应的多肽都符合已知基序。在非靶向质谱的结果中,观察到22个新抗原-HLA结合表位,与5个HLA分型中的15个共有新生抗原相对应,并且这22个表位在TR-FRET和NetMHC中的预测一致性非常高,其中有17个在两种预测方法中都被认定为结合位点。
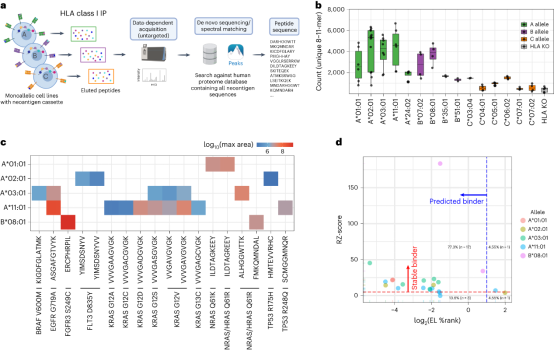
图3 工程T细胞免疫多肽组的质谱鉴定
4. PRM靶向验证候选结合表位
首先通过TR-FRET RZ评分≥5初步筛选出397条多肽,然后结合NetMHC %Rank≤2和RZ评分< 5筛选出互补的81条,共计对479条候选的新抗原多肽进行合成,然后通过PRM进行靶向鉴定。结果共筛选出86个新抗原表位-HLA结合对和36条新生抗原肽。86个结合对中,有21个是已发表过的,65个是未曾报道的新发现,共55个被TR-FRET与NetMHC预测均认定为预测结合位点。另外,通过与非靶向质谱数据相比,靶向法能更好地识别出结合能力较弱的新表位,且能实现绝对定量。
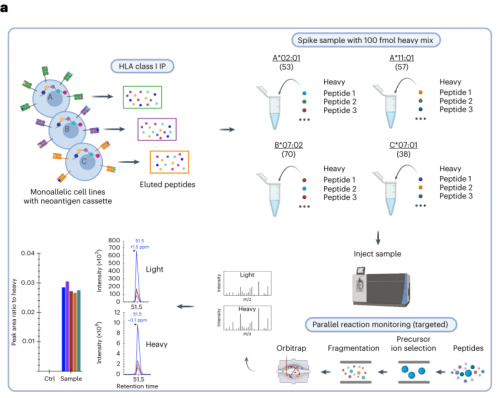
图4a 靶向质谱法验证工程T细胞的免疫多肽组
5. 全长蛋白的KRAS新抗原表位验证
为了验证由全长突变蛋白加工生成的新生抗原肽,构建了4个HLA-A*11:01单等位基因突变的C1R细胞系,包括全长野生型、G12C、G12D或G12V突变的KRAS,并通过全细胞靶向蛋白组验证各自的信号肽。然后,将先前鉴定到的与HLA-1*11:01相关的两个KRAS表位进行富集和靶向定量。与全长野生型细胞系相比,G12V和G12D的均被明显诱导产生更多VVVGAVGVGK的结合表位。最后,在内源性表达相关蛋白和HLA的细胞系中,进一步验证了发现的候选新抗原结合表位。
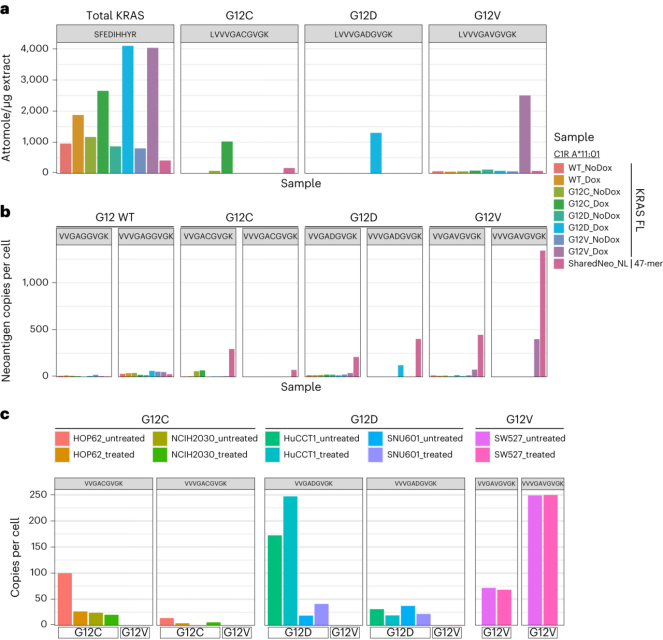
图5 全长蛋白新抗原表位验证
6. 肿瘤特异性抗原-HLA结合对的功能验证
采用了复合TCR发现法进一步验证了通过该方案筛选出的新抗原表位可以被人的T细胞识别。以鉴定到的两个新表位-HLA结合对(FLT3-p.D835Y/A*02:01, PIK3CA-p.E545K/A*11:01))为例,首先将新表位以独特的组合分配到多肽库中,然后使用自体单核细胞来源的树突细胞对健康人供体CD8+ T细胞进行扩增,接着用新表位肽库重新激活T细胞,再通过激活标志物排序,最后进行TCRβ测序。结果发现,与野生型相比,PIK3CA-p.E545K TCR能够特异性识别突变的PIK3CA,并且有可能是潜在的治疗靶点。
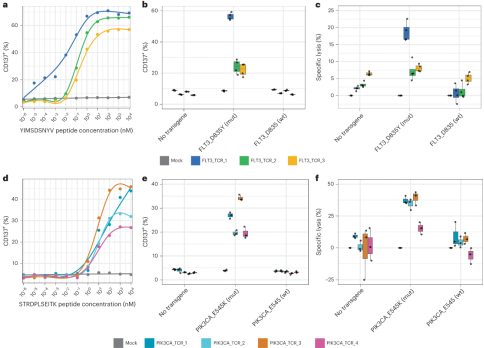
图6 筛查新抗原-HLA结合对的特异性TCR以验证免疫原性
综上所述,本研究创建了一个可扩展的新抗原表位- HLA结合对的发现策略。首先选择了47个癌症突变和15个HLA等位基因来定义新抗原表位,并由此扩展到假定的临床靶点。然后,采用高通量HLA结合测定法5.6和NetMHCpan-4.07,在湿实验和计算预测两个方面对新抗原-HLA结合对进行筛选。针对上述方法观察到的新抗原-HLA结合对,分别采用非靶向和靶向质谱法(MS)对HLA单等位细胞系的免疫多肽组进行分析,最终得到86个新抗原-HLA结合对。为了评估这些靶点的治疗潜力,通过TCR证明了突变选择性T细胞对有这些新抗原表达的细胞的靶向杀伤力。
原文链接:https://doi.org/10.1038/s41587-023-01945-y
目前在PEAKS的软件中,针对多肽组分析,尤其是以免疫多肽为代表的应用场景,我们已经推出基于深度学习技术的DeepNovo Peptidome工作流,原有PEAKS de novo+ PEAKS DB的分析基础上,通过氨基酸水平的置信度、保留时间和CCS值等预测,特别是巧妙地通过两轮分析将从头测序的多肽结果纳入全局FDR的质控,对于多肽组鉴定的灵敏度和准确度进一步提高。
参考文献:
1. Jhunjhunwala, S., Hammer, C. & Delamarre, L. Antigen presentation in cancer: insights into tumour immunogenicity and immune evasion. Nat. Rev. Cancer 21, 298–312 (2021).
2. Klebanoff, C. A. & Wolchok, J. D. Shared cancer neoantigens: making private matters public. J. Exp. Med. 215, 5–7 (2018).
3. Zhang, Z. et al. Neoantigen: a new breakthrough in tumor immunotherapy. Front. Immunol. 12, 672356 (2021).
4. Mei, S. et al. A comprehensive review and performance evaluation of bioinformatics tools for HLA class I peptide-binding prediction. Brief. Bioinform. 21, 1119–1135 (2020).
5. Darwish, M. et al. High‐throughput identification of conditional MHCI ligands and scaled‐up production of conditional MHCI complexes. Protein Sci. 30, 1169–1183 (2021).
6. Rodenko, B. et al. Generation of peptide–MHC class I complexes through UV-mediated ligand exchange. Nat. Protoc. 1, 1120–1132 (2006).
7. Jurtz, V. et al. NetMHCpan-4.0: improved peptide–MHC class I interaction predictions integrating eluted ligand and peptide binding affinity data. J. Immunol. 199, 3360–3368 (2017).
8. https://www.bioinfor.com/deepnovo-peptidome/